automap::autoKrige fit.method 의존성 문제
공간 보간 기법 중 하나인 Kriging 을 R에서는 사용하기 편리하기 위해서 automap패키지에서 autoKrige로 지원하고 있다. 하지만 최근 fitting 과정에서 fit.method가 gstats 패키지에 있어 옵션이 수정되지 않는 현상을 겪었다.
따라서 아래와같이 function을 올려보겠다.
autoKrige2=function (formula, input_data, new_data, data_variogram = input_data,
block = 0, model = c("Sph", "Exp", "Gau", "Ste"), kappa = c(0.05,
seq(0.2, 2, 0.1), 5, 10), fix.values = c(NA, NA, NA),
remove_duplicates = TRUE, verbose = FALSE, GLS.model = NA, fit.method=fit.method,
start_vals = c(NA, NA, NA), miscFitOptions = list(), ...)
{
if (inherits(formula, "SpatialPointsDataFrame")) {
input_data = formula
formula = as.formula(paste(names(input_data)[1], "~ 1"))
}
if (!inherits(input_data, "SpatialPointsDataFrame") | !inherits(data_variogram,
"SpatialPointsDataFrame")) {
stop(paste("\nInvalid input objects: input_data or data_variogram not of class 'SpatialPointsDataFrame'.\n\tClass input_data: '",
class(input_data), "'", "\n\tClass data_variogram: '",
class(data_variogram), "'", sep = ""))
}
if (as.character(formula)[3] != 1 & missing(new_data))
stop("If you want to use Universal Kriging, new_data needs to be specified \n because the predictors are also required on the prediction locations.")
if ("newdata" %in% names(list(...)))
stop("The argument name for the prediction object is not 'newdata', but 'new_data'.")
if (remove_duplicates) {
zd = zerodist(input_data)
if (length(zd) != 0) {
warning("Removed ", length(zd)/2, " duplicate observation(s) in input_data:",
immediate. = TRUE)
print(input_data[c(zd), ])
input_data = input_data[-zd[, 2], ]
}
}
col_name = as.character(formula)[2]
if (length(unique(input_data[[col_name]])) == 1)
stop(sprintf("All data in attribute '%s' is identical and equal to %s\n Can not interpolate this data",
col_name, unique(input_data[[col_name]])[1]))
if (missing(new_data))
new_data = create_new_data(input_data)
p4s_obj1 = proj4string(input_data)
p4s_obj2 = proj4string(new_data)
if (!all(is.na(c(p4s_obj1, p4s_obj2)))) {
if (is.na(p4s_obj1) & !is.na(p4s_obj2))
proj4string(input_data) = proj4string(new_data)
if (!is.na(p4s_obj1) & is.na(p4s_obj2))
proj4string(new_data) = proj4string(input_data)
if (any(!c(is.projected(input_data), is.projected(new_data))))
stop(paste("Either input_data or new_data is in LongLat, please reproject.\n",
" input_data: ", p4s_obj1, "\n", " new_data: ",
p4s_obj2, "\n"))
if (proj4string(input_data) != proj4string(new_data))
stop(paste("Projections of input_data and new_data do not match:\n",
" input_data: ", p4s_obj1, "\n", " new_data: ",
p4s_obj2, "\n"))
}
variogram_object = autofitVariogram2(formula, data_variogram,
model = model, kappa = kappa, fix.values = fix.values,
verbose = verbose, GLS.model = GLS.model, start_vals = start_vals,
miscFitOptions = miscFitOptions, fit.method =fit.method)
krige_result = krige(formula, input_data, new_data, variogram_object$var_model,
block = block, ...)
krige_result$var1.stdev = sqrt(krige_result$var1.var)
result = list(krige_output = krige_result, exp_var = variogram_object$exp_var,
var_model = variogram_object$var_model, sserr = variogram_object$sserr)
class(result) = c("autoKrige", "list")
return(result)
}
autofitVariogram2=function (formula, input_data, model = c("Sph", "Exp", "Gau",
"Ste"), kappa = c(0.05, seq(0.2, 2, 0.1), 5, 10), fix.values = c(NA,
NA, NA), verbose = FALSE, GLS.model = NA, start_vals = c(NA,
NA, NA), miscFitOptions = list(),fit.method = 1, ...)
{
if ("alpha" %in% names(list(...)))
warning("Anisotropic variogram model fitting not supported, see the documentation of autofitVariogram for more details.")
miscFitOptionsDefaults = list(merge.small.bins = TRUE, min.np.bin = 5)
miscFitOptions = modifyList(miscFitOptionsDefaults, miscFitOptions)
longlat = !is.projected(input_data)
if (is.na(longlat))
longlat = FALSE
diagonal = spDists(t(bbox(input_data)), longlat = longlat)[1,
2]
boundaries = c(2, 4, 6, 9, 12, 15, 25, 35, 50, 65, 80, 100) *
diagonal * 0.35/100
if (!is(GLS.model, "variogramModel")) {
experimental_variogram = variogram(formula, input_data,
boundaries = boundaries, ...)
}
else {
if (verbose)
cat("Calculating GLS sample variogram\n")
g = gstat(NULL, "bla", formula, input_data, model = GLS.model,
set = list(gls = 1))
experimental_variogram = variogram(g, boundaries = boundaries,
...)
}
if (miscFitOptions[["merge.small.bins"]]) {
if (verbose)
cat("Checking if any bins have less than 5 points, merging bins when necessary...\n\n")
while (TRUE) {
if (length(experimental_variogram$np[experimental_variogram$np <
miscFitOptions[["min.np.bin"]]]) == 0 | length(boundaries) ==
1)
break
boundaries = boundaries[2:length(boundaries)]
if (!is(GLS.model, "variogramModel")) {
experimental_variogram = variogram(formula,
input_data, boundaries = boundaries, ...)
}
else {
experimental_variogram = variogram(g, boundaries = boundaries,
...)
}
}
}
if (is.na(start_vals[1])) {
initial_nugget = min(experimental_variogram$gamma)
}
else {
initial_nugget = start_vals[1]
}
if (is.na(start_vals[2])) {
initial_range = 0.1 * diagonal
}
else {
initial_range = start_vals[2]
}
if (is.na(start_vals[3])) {
initial_sill = mean(c(max(experimental_variogram$gamma),
median(experimental_variogram$gamma)))
}
else {
initial_sill = start_vals[3]
}
if (!is.na(fix.values[1])) {
fit_nugget = FALSE
initial_nugget = fix.values[1]
}
else fit_nugget = TRUE
if (!is.na(fix.values[2])) {
fit_range = FALSE
initial_range = fix.values[2]
}
else fit_range = TRUE
if (!is.na(fix.values[3])) {
fit_sill = FALSE
initial_sill = fix.values[3]
}
else fit_sill = TRUE
getModel = function(psill, model, range, kappa, nugget,
fit_range, fit_sill, fit_nugget, verbose) {
if (verbose)
debug.level = 1
else debug.level = 0
if (model == "Pow") {
warning("Using the power model is at your own risk, read the docs of autofitVariogram for more details.")
if (is.na(start_vals[1]))
nugget = 0
if (is.na(start_vals[2]))
range = 1
if (is.na(start_vals[3]))
sill = 1
}
obj = try(gstat::fit.variogram(experimental_variogram, model = vgm(psill = psill,
model = model, range = range, nugget = nugget, kappa = kappa),
fit.ranges = c(fit_range), fit.sills = c(fit_nugget,
fit_sill), debug.level = 0,fit.method = fit.method), TRUE)
if ("try-error" %in% class(obj)) {
warning("An error has occured during variogram fitting. Used:\n",
"\tnugget:\t", nugget, "\n\tmodel:\t", model,
"\n\tpsill:\t", psill, "\n\trange:\t", range,
"\n\tkappa:\t", ifelse(kappa == 0, NA, kappa),
"\n as initial guess. This particular variogram fit is not taken into account. \nGstat error:\n",
obj)
return(NULL)
}
else return(obj)
}
test_models = model
SSerr_list = c()
vgm_list = list()
counter = 1
for (m in test_models) {
if (m != "Mat" && m != "Ste") {
model_fit = getModel(initial_sill - initial_nugget,
m, initial_range, kappa = 0, initial_nugget,
fit_range, fit_sill, fit_nugget, verbose = verbose)
if (!is.null(model_fit)) {
vgm_list[[counter]] = model_fit
SSerr_list = c(SSerr_list, attr(model_fit, "SSErr"))
}
counter = counter + 1
}
else {
for (k in kappa) {
model_fit = getModel(initial_sill - initial_nugget,
m, initial_range, k, initial_nugget, fit_range,
fit_sill, fit_nugget, verbose = verbose)
if (!is.null(model_fit)) {
vgm_list[[counter]] = model_fit
SSerr_list = c(SSerr_list, attr(model_fit,
"SSErr"))
}
counter = counter + 1
}
}
}
strange_entries = sapply(vgm_list, function(v) any(c(v$psill,
v$range) < 0) | is.null(v))
if (any(strange_entries)) {
if (verbose) {
print(vgm_list[strange_entries])
cat("^^^ ABOVE MODELS WERE REMOVED ^^^\n\n")
}
warning("Some models where removed for being either NULL or having a negative sill/range/nugget, \n\tset verbose == TRUE for more information")
SSerr_list = SSerr_list[!strange_entries]
vgm_list = vgm_list[!strange_entries]
}
if (verbose) {
cat("Selected:\n")
print(vgm_list[[which.min(SSerr_list)]])
cat("\nTested models, best first:\n")
tested = data.frame(`Tested models` = sapply(vgm_list,
function(x) as.character(x[2, 1])), kappa = sapply(vgm_list,
function(x) as.character(x[2, 4])), SSerror = SSerr_list)
tested = tested[order(tested$SSerror), ]
print(tested)
}
result = list(exp_var = experimental_variogram, var_model = vgm_list[[which.min(SSerr_list)]],
sserr = min(SSerr_list))
class(result) = c("autofitVariogram", "list")
return(result)
}'R' 카테고리의 다른 글
| 딥로또 R 버전 (0) | 2020.02.10 |
|---|---|
| R 스케줄링 (0) | 2020.01.19 |
| [ggplot2] 시각화 정리 (0) | 2019.12.08 |
| [R][SQL] RMariaDB 외부 접속 설정하기 (0) | 2019.11.29 |
| [공간정보오픈플렛폼]위경도 변환, 주소 변환 (0) | 2019.11.27 |
얼마전에 로또분석에 관해 김태영님께서 포스팅한 딥러닝 글을 보게되었습니다. 평소에 로또나 주식에 관심이 많았기에 기존 분석방법을 고도화 하고 싶어 김태영님의 코드를 참고하였고, 이를 R버전으로 포스팅해보겠다. 물론 현재 이 외에 다양한 시도를 해보는 중이다.
참고 : 김태영님의 블로그
딥로또 895회
딥러닝 세미나를 하다보면 (특히 RNN이나 LSTM 등 시계열 예측 모델을 설명하다보면) 로또나 주식에 관해 질문을 많이 하십니다. 이와 관련된 질문에는 나름 원칙이 있거나 시계열적 특성이나 인과관계가 있는 데이터에만 예측이 잘될꺼라도 말씀은 드리긴 하지만 신통치 않은 답변인지 만족하시질 못 합니다. 그래서 먼저 딥러닝으로 로또를 하면 안된다는 것을 몸소 보여드리기 위해 매주 테스트를 해볼 예정입니다. 들어가기에 앞서 몇가지 말씀드리겠습니다. 로또 같은
tykimos.github.io
로또 자료 생성
로또 자료 생성의 경우 멀티 코어를 활용하였다. 또한 패키지 로드가 귀찮아 만들었던 개인 패키지를 활용하였다.
library(devtools)
if(!require(lotto))install_github('qkdrk777777/lotto')
if(!require(DUcj))install_github('qkdrk777777/DUcj')
# 개인 패키지
library(DUcj)
# DUcj::package 는 if(!require(패키지))install.packages('패키지')를 동시에 진행해주는 함수
package('progress')
package('XML')
package('stringr')
package('rvest')
package(parallel)
package(foreach)
package(rvest)
package(xml2)
url<-'https://search.naver.com/search.naver?sm=tab_hty.top&where=nexearch&query=%EB%A1%9C%EB%98%90&oquery=%EB%A1%9C%EB%98%90&tqi=TmrKidpVuFdsssc0EvVssssssUd-075322'
line<-read_html(url,encoding="UTF-8")
p1<-html_nodes(line,css='._lotto-btn-current em')%>%html_text()
last=as.numeric(substr(p1,1,3))
last
spec=detectCores()
spec
cl=makeCluster(spec)
data=NULL
try({url<-paste0('https://search.naver.com/search.naver?sm=tab_drt&where=nexearch&query=',last:1,'%ED%9A%8C%EB%A1%9C%EB%98%90')
c=data.frame(t(parApply(cl,MARGIN=1,data.frame(url),function(x){
(as.numeric(strsplit(stringr::str_trim(
rvest::html_text(rvest::html_nodes(xml2::read_html(x,encoding='UTF-8'),css='.num_box'))),split=' ')[[1]][1:6]))})))
data=cbind(num=last:1,c)},silent = T)
stopCluster(cl)
Sys.sleep(3)
# 멀티코어로 크롤링을 실패한 경우 다음과 같이 진행
if(is.null(data)){
url<-paste0('https://search.naver.com/search.naver?sm=tab_drt&where=nexearch&query=',last:1,'%ED%9A%8C%EB%A1%9C%EB%98%90')
data=NULL;t=0
for( i in url){t=t+1
data=rbind(data,data.frame(num=(last:1)[t],t(as.numeric(strsplit(stringr::str_trim(
rvest::html_text(rvest::html_nodes(xml2::read_html(i,encoding = 'UTF-8'),css='.num_box'))),split=' ')[[1]][1:6]))))
message(t/last*100)
}
}
분석용 데이터 생성
library(plyr)
rm(list=setdiff(ls(),c('data','data2')))
data2<-matrix(0,ncol=45,nrow=nrow(data));colnames(data2)=1:45
for(i in 1:nrow(data2)){
for(j in 2:7){
for(k in 1:45){
if(data[i,j]==k)data2[i,k]<-1}}}
rownames(data2)<-nrow(data2):1
library(keras)
library(tensorflow)
library(reticulate)
delete=data2
train=data.matrix(delete[-1,])
train=array(train,dim=c(1,nrow(train),ncol(train)))
target=data.matrix(delete[-nrow(data),])
모델 생성
model=keras_model_sequential()%>%
layer_cudnn_lstm(units=128,batch_input_shape=c(1,1,dim(train)[3]),return_sequences = F,stateful = T)%>%
layer_dense(units=45,activation = 'sigmoid')
model %>% compile(
optimizer = "adam",
loss = 'binary_crossentropy')
M_train_loss=c()
M_val_loss=c()
for(epoch in 1:150){
model$reset_states()
train_loss=c()
for (i in 1:700){
xs=train[,i,,drop=F]
ys=target[i,,drop=F]
train_loss=c(loss,model$train_on_batch(xs,ys))}
val_loss=c()
for( i in 700:800){
xs=train[,i,,drop=F]
ys=target[i,,drop=F]
val_loss=c(loss,model$test_on_batch(xs,ys))}
M_train_loss=c(M_train_loss,mean(train_loss))
M_val_loss=c(M_val_loss,mean(val_loss))
print(epoch)
}
matplot(cbind(M_train_loss,M_val_loss),type='l')
ls=list()
for(i in 1:800){
source('./MC_test.R')
}
ls1=ls
ls=list()
for(i in (800:(nrow(data)-1))){
source('./MC_test.R')
}
ls2=ls
table(rep(names(unlist(ls1)),unlist(ls1)))
table(rep(names(unlist(ls2)),unlist(ls2)))
i=dim(train)[2]
ball_list=c()
for(j in 1:10){
xs=train[,i,,drop=F]
pred=model$predict_on_batch(xs)
ball_box=rep(1:45,as.integer(pred*100+1))
selected_balls=c()
while(T){
if (length(selected_balls)==6) break
ball=sample(ball_box,1)
if (!(ball %in% selected_balls)) selected_balls=c(selected_balls,ball)
}
ball_list=rbind(ball_list,sort(selected_balls))
}
source 코드로 사용된 MC_test.R 코드
검증 코드는 편의상 2등을 생략하였는데 크롤링으로 데이터 생성할 때부터 보너스번호를 고려하여 짜면 될거 같다.
rank=c()
for(j in 1:10){
xs=train[,i,,drop=F]
pred=model$predict_on_batch(xs)
ball_box=rep(1:45,as.integer(pred*100+1))
selected_balls=c()
while(T){
if (length(selected_balls)==6) break
ball=sample(ball_box,1)
if (!(ball %in% selected_balls)) selected_balls=c(selected_balls,ball)
}
selected_balls
temp=length(intersect(selected_balls,data[i+1,-1]))
rank=c(rank,ifelse(temp==6,'1등',ifelse(temp==5,'3등',ifelse(temp==4,'4등',ifelse(temp==3,'5등',NA)))))
}
# print(i)
ls[[i]]=table(rank)
참고 : loss function 을 커스터마이징하여 분석을 진행할때 활용한 코드
lossfunction <- function(y, t) {
k_binary_crossentropy(y,t)
}
model %>% compile(
optimizer = "adam",
loss = function(y_true, y_pred)
lossfunction(y_true, y_pred)
)
'R' 카테고리의 다른 글
| automap::autoKrige fit.method 의존성 문제 (0) | 2020.07.18 |
|---|---|
| R 스케줄링 (0) | 2020.01.19 |
| [ggplot2] 시각화 정리 (0) | 2019.12.08 |
| [R][SQL] RMariaDB 외부 접속 설정하기 (0) | 2019.11.29 |
| [공간정보오픈플렛폼]위경도 변환, 주소 변환 (0) | 2019.11.27 |
rstudio server 관리 및 설정
rstudio server 관리 파일의 구성
rstudio server 의 구성파일의 경우 /etc/rstudio/ 내에 존재
기본적으로 rserver.conf와 rsession.conf가 존재하며 나머지는 만들어 사용 가능

서버 실행 및 중지 관련 코드
- sudo rstudio-server stop
- sudo rstudio-server start
- sudo rstudio-server restart
서버 실행 상태확인 코드
- sudo rstudio-server status
사용중인 계정 확인 코드
- sudo rstudio-server active-sessions
새 버전의 Rstudio server 업그레이드
- sudo gdebi <rstudio-server-package.deb>
example)
wget https://download2.rstudio.org/server/bionic/amd64/rstudio-server-1.2.5033-amd64.deb sudo gdebi rstudio-server-1.2.5033-amd64.deb
rserver.conf 옵션
접속 가능 사용자 목록 관리
- auth-required-user-group=유저명
포트 번호 수정
- www-port=80
외부 아이피 허용
- www-address=127.0.0.1
아이피 접속 허용 및 차단
/ etc / rstudio / ip-rules
example) 192.168.1.0/24 만 허용 나머지 차단
deny 192.168.1.10
allow 192.168.1.0/24
deny all
r-studio server 실행될 때 먼저 실행되는 스크립트 조절하기.
/etc/rstudio/rsession-profile
프로파일 생성
/etc/rstudio/profiles
1.글로벌( [*])
2.그룹([@그룹명])
3.사용자([유저명])
example)
[*] cpu-affinity = 1-4
max-processes = 100
max-memory-mb = 2048
session-timeout-minutes=60
[@powerusers] cpu-affinity = 5-16
nice = -10
max-memory-mb = 4096
[jsmith] r-version = /opt/R/3.1.0
session-timeout-minutes=360
rsession.conf 옵션
세션 타임아웃
- 세션이 일시중단 되기 전에 대기할 시간을 설정
- 기본 120분
- session-timeout-minutes=0으로 세션이 일시중단되지 않게 설정 가능
스케줄링 우선순위
- nice 를 조절하여 상대적 우선순위 설정 기본값은 0이며 -20~20까지 설정이 가능 -20으로 갈수록 우선순위가 높아짐.
메모리 제한 설정
max-memory-mb=제한할 메모리 량
기본값은 무제한 최소 1024 이상은 설정해야 됨
버전 관리
r-version 옵션을 통해 사용자별 r-version 관리 가능
[참고]
https://docs.rstudio.com/ide/server-pro/1.0.153/authenticating-users.html
'R > Rstudio Server' 카테고리의 다른 글
| 우분투에서 R로 Selenium 사용하기(도커 사용) (0) | 2019.03.23 |
|---|---|
| R 패키지 설치가 안될 때(RCurl, devtools, rvest) 및 Rstudio 서버 사용자 추가 (0) | 2019.03.15 |
| ubuntu18.04 r-studio server setup (0) | 2019.03.15 |
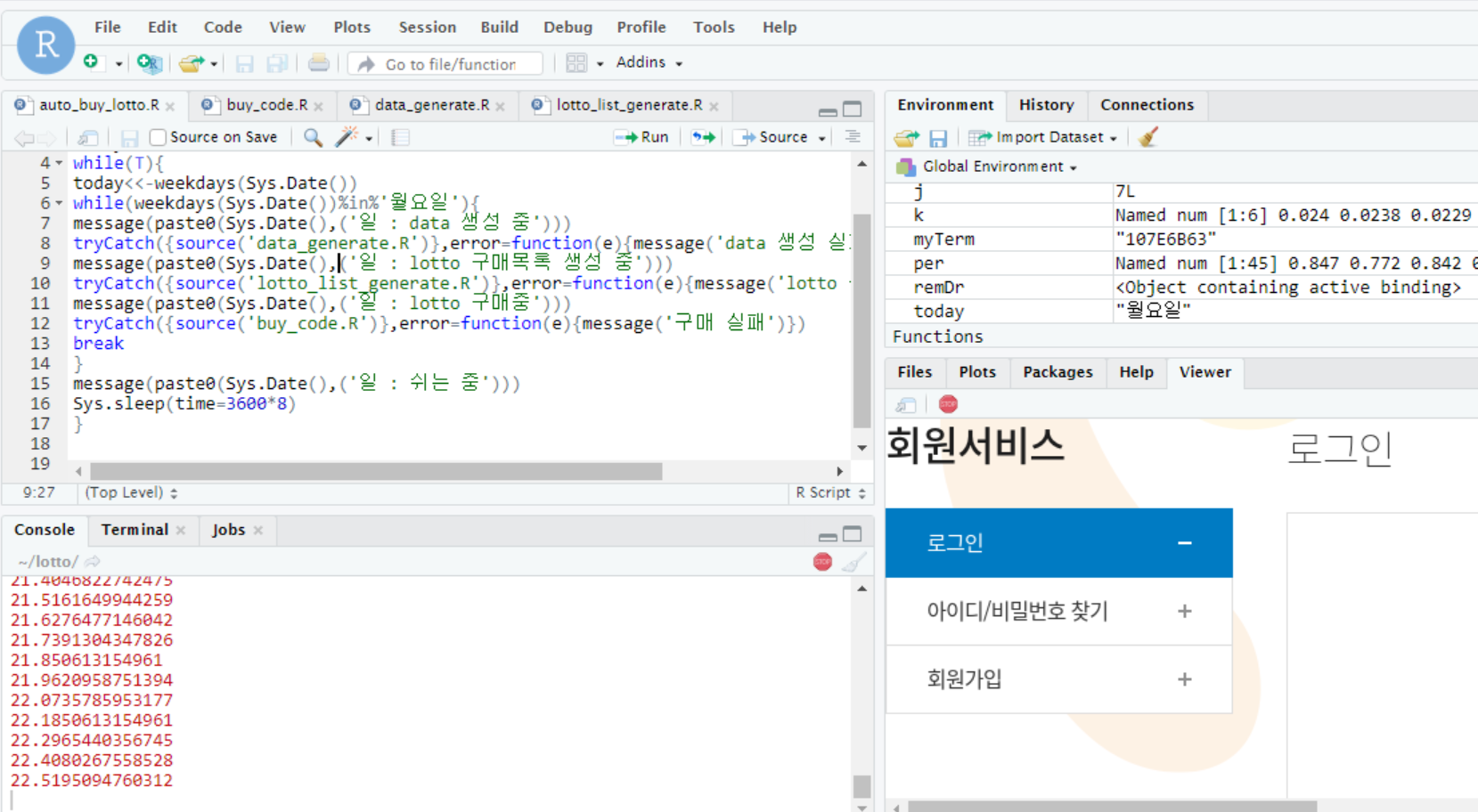
특정 기간마다 스크립트 실행
패키지 설치 후 최종적으로 보이는 화면은 아래와 같다.
browse...를 통해 스크립트를 고를 수 있으며,
Rscript repo : location where Rscripts will be copied to schedule + location of logs 를 통해 스케줄 및 로그파일의 저장경로를 성정 가능하다.
stop or Delete를 통해 스케줄을 제거 가능하다.
스크립트가 한글 지원을 하지 않는 점을 참고하기 바란다.

우분투18.04에서는?
sudo apt-get update
sudo apt-get install -y cron
sudo cron start
devtools::install_github("bnosac/cronR")
install.packages('miniUI')
install.packages('shiny')
install.packages('shinyFiles')

'R' 카테고리의 다른 글
| automap::autoKrige fit.method 의존성 문제 (0) | 2020.07.18 |
|---|---|
| 딥로또 R 버전 (0) | 2020.02.10 |
| [ggplot2] 시각화 정리 (0) | 2019.12.08 |
| [R][SQL] RMariaDB 외부 접속 설정하기 (0) | 2019.11.29 |
| [공간정보오픈플렛폼]위경도 변환, 주소 변환 (0) | 2019.11.27 |
[ggplot2] 시각화 정리
ggplot(data, aes(x축, y축, fill=범주))
geom_bar : 바 그래프
geom_point : 산점도
geom_line : 라인 그래프
geom_count: 버블차트
geom_label: 라벨 입력
geom_errorbar: 에러 바
ggtitle : 그림제목
coord_flip : 가로 막대그래프로 변경
theme(panel.background = element_rect(fill= "transparent"))
theme_classic()
facet_grid, facet_wrap : 면 분할
xlim : x축의 범위
ylim : y축의 범위
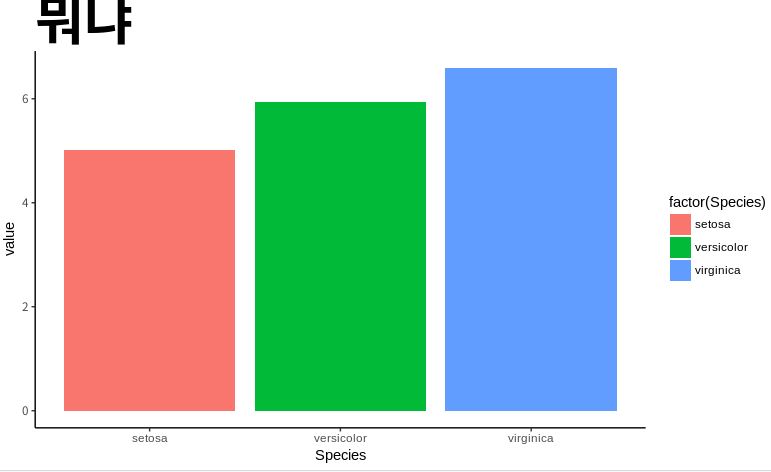
예제1
data(iris)
del=data.table::setDT(iris)[,.(mean=mean(Sepal.Length)),'Species']
del=reshape2::melt(del,'Species')
library(ggplot2)
ggplot(del,aes(Species,value,fill=factor(Species)))+geom_bar(stat='identity')+
theme_classic()+theme(plot.title=element_text(face='bold',size=40),
legend.box.just = 'right',legend.margin = margin(6,6,6,6))+
labs(fill='Species')+ggtitle('뭐냐')
예제2
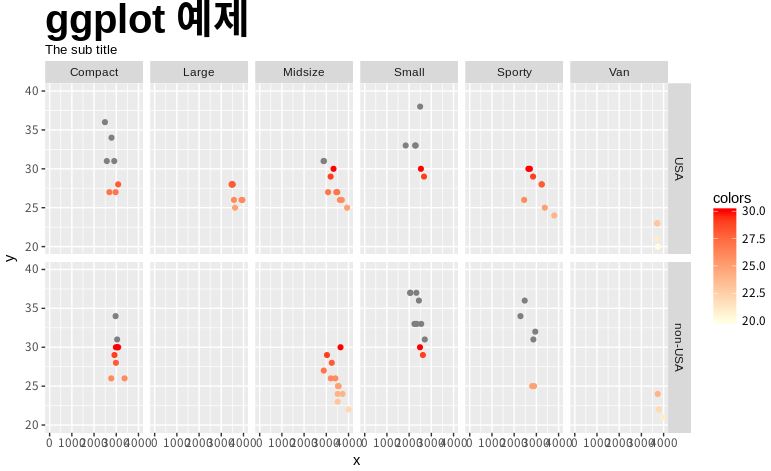
ggplot(Cars93, aes(x=Weight, y=MPG.highway,colour=MPG.highway)) +
geom_point() +scale_colour_gradient(low='lightyellow', high='red',limits=c(20,30))+
facet_grid(Origin ~ Type)+ggtitle('ggplot 예제')+theme(plot.title=element_text(face='bold',size=30))+
labs(
subtitle = 'The sub title',
x = 'x', y = 'y', color = 'colors')+xlim(c(0,4000))+ylim(c(20,40))
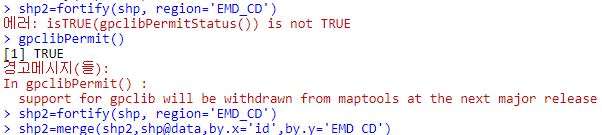
library(rgdal)
shp=readOGR('C:/Users/USER/Desktop/CJ/shp/CTPRVN_201703/TL_SCCO_CTPRVN.shp',encoding = 'UTF-8')
crs=proj4string(shp)
shp=spTransform(shp,CRS('+init=epsg:4326'))
set.seed(1)
temp=data.frame(id=shp@data$CTP_KOR_NM,random=sample(1:1000,length(shp@data$CTP_KOR_NM)),stringsAsFactors = F)
library(ggmap)
head(shp@data)
shp2=fortify(shp,region='CTP_KOR_NM')
shp3=dplyr::left_join(shp2,temp,'id')
register_google(key=키)
location=geocode('대한민국')
map=get_googlemap(center = unlist(location),zoom=5)
ggmap(map)+geom_polygon(data=shp3,aes(x=long,y=lat,group=group,fill=random))+scale_fill_gradient(limits=c(250,750)) 
bar plot 의 xlim이 0보다 크게 시작할 때 coord_cartesian(ylim = c(.6, 1),xlim=c(0,24))를 써 해결하면 된다.
'R' 카테고리의 다른 글
| 딥로또 R 버전 (0) | 2020.02.10 |
|---|---|
| R 스케줄링 (0) | 2020.01.19 |
| [R][SQL] RMariaDB 외부 접속 설정하기 (0) | 2019.11.29 |
| [공간정보오픈플렛폼]위경도 변환, 주소 변환 (0) | 2019.11.27 |
| [ggmap] pie chart 그리기 예제 (0) | 2019.11.26 |
[R][SQL] RMariaDB 외부 접속 설정하기
ubuntu18.04에서 RMariaDB를 설치부분은 생략하고 외부접속 설정 부분만을 언급하겠다. 외부 접속 설정을 위해서는 다음과 같은 다양한 절차가 필요하다.
방화벽 해제
먼저 포트포워딩을 통해 포트를 5800, 5900, 3306을 열어준다.

터미널에서 아래와 같이 명령어로 3306, 5900, 5800 포트를 개방해준다.
sudo ufw allow 3306
sudo ufw allow 5900
sudo ufw allow 5800
mariaDB의 외부접속 허용
/etc/mysql/mariadb.conf.d 경로의 50-server.cnf에서 bind-address 를 0.0.0.0으로 변경해준다.


MariaDB에서 개정을 만들 때 create user '[username]'@'[host]' 부분에서 host는 외부 ip를 입력하면 된다.
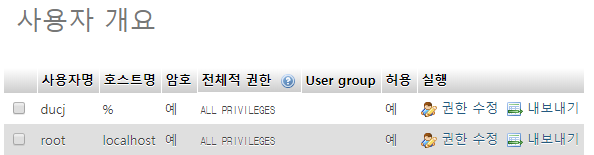
#MariaDB 접속
mysql -u root -p
#계정 생성
create user '[username]'@'[host or %]' identified by '[password]';
#권한 부여
grant all privileges on [database].* to '[username]'@'[host]';
use mysql;
#유저 목혹 확인
select user, host from mysql.user;
#MariaDB 권한초기화
flush privileges;
#MariaDB 재시작
sudo service mysql restart
#MariaDB 유저 제거
delete from user wher user='[username]';
flush privileges;R에서의 적용

'R' 카테고리의 다른 글
| R 스케줄링 (0) | 2020.01.19 |
|---|---|
| [ggplot2] 시각화 정리 (0) | 2019.12.08 |
| [공간정보오픈플렛폼]위경도 변환, 주소 변환 (0) | 2019.11.27 |
| [ggmap] pie chart 그리기 예제 (0) | 2019.11.26 |
| ddply와 setDT 비교 (0) | 2019.11.11 |
